gitのコマンドの出力を色分けして表示する
$ git config --global color.ui "auto" $ git config --global core.pager 'less -R'
マニュアルを読む限りでは1行目だけでいけそうなんだけど,僕の環境では2行目がないとlessでエスケープシーケンスが化けました.
Purely Functional Data Structures 第2章
演習問題を解いたのでメモ.
ex 2.1
suffixes :: [a] -> [[a]] suffixes [] = [[]] suffixes xss@(_:xs) = xss : suffixes xs
ex 2.2
data Set a = Leaf | Branch a (Set a) (Set a) deriving Show empty :: Set a empty = Leaf insert :: Ord a => a -> Set a -> Set a insert x Leaf = Branch x Leaf Leaf insert x tree@(Branch y left right) | x < y = Branch y (insert x left) right | x > y = Branch y left (insert x right) | otherwise = tree member :: Ord a => a -> Set a -> Bool member x set = member' Nothing set where member' c Leaf = maybe False (>= x) c member' c (Branch y left right) | x < y = member' c left | otherwise = member' (Just y) right
ex 2.3
「Establish only one handler per insertion rather than one handler per iteration」の意味がわからなかったので,この文は無視して解いた.
import Data.Maybe data Set a = Leaf | Branch a (Set a) (Set a) deriving Show empty :: Set a empty = Leaf insert :: Ord a => a -> Set a -> Set a insert x set = fromMaybe set (insert' set) where insert' Leaf = Just (Branch x Leaf Leaf) insert' (Branch y left right) | x < y = fmap (\new -> Branch y new right) (insert' left) | x > y = fmap (\new -> Branch y left new) (insert' right) | otherwise = Nothing member :: Ord a => a -> Set a -> Bool member x set = member' Nothing set where member' c Leaf = maybe False (>= x) c member' c (Branch y left right) | x < y = member' c left | otherwise = member' (Just y) right
ex 2.4
import Data.Maybe data Set a = Leaf | Branch a (Set a) (Set a) deriving Show empty :: Set a empty = Leaf insert :: Ord a => a -> Set a -> Set a insert x set = fromMaybe set (insert' Nothing set) where insert' c Leaf | isNothing c || x > fromJust c = Just (Branch x Leaf Leaf) | otherwise = Nothing insert' c (Branch y left right) | x < y = fmap (\new -> Branch y new right) (insert' c left) | otherwise = fmap (\new -> Branch y left new) (insert' (Just y) right) member :: Ord a => a -> Set a -> Bool member x set = member' Nothing set where member' c Leaf = maybe False (>= x) c member' c (Branch y left right) | x < y = member' c left | otherwise = member' (Just y) right
ex 2.5
data Tree a = Empty | Node a (Tree a) (Tree a) deriving Show makeCompleteBinaryTree :: a -> Int -> Tree a makeCompleteBinaryTree _ 0 = Empty makeCompleteBinaryTree x d = let subtree = makeCompleteBinaryTree x (d-1) in Node x subtree subtree makeBinaryTree :: a -> Int -> Tree a makeBinaryTree x n = fst (make2 n) where make2 0 = (Empty, Node x Empty Empty) make2 n | n `mod` 2 == 0 = let (t1, t2) = make2 (n `div` 2 - 1) in (Node x t1 t2, Node x t2 t2) | otherwise = let (t1, t2) = make2 (n `div` 2) in (Node x t1 t1, Node x t1 t2)
ex 2.6
import Data.Maybe data FinateMap k v = Leaf | Branch k v (FinateMap k v) (FinateMap k v) deriving Show empty :: FinateMap k v empty = Leaf bind :: Ord k => k -> v -> FinateMap k v -> FinateMap k v bind k v m = fromMaybe m (bind' Nothing m) where bind' c Leaf | isNothing c || k > fromJust c = Just (Branch k v Leaf Leaf) | otherwise = Nothing bind' c (Branch k' v' left right) | k < k' = fmap (\new -> Branch k' v' new right) (bind' c left) | otherwise = fmap (\new -> Branch k' v' left new) (bind' (Just k') right) lookup :: Ord k => k -> FinateMap k v -> Maybe v lookup k m = lookup' Nothing m where lookup' (Just (k', v')) Leaf | k' >= k = Just v' lookup' c (Branch k' v' left right) | k < k' = lookup' c left | otherwise = lookup' (Just (k', v')) right lookup' _ _ = Nothing
ICPC国内予選2011に参加しました
ICPC国内予選にチーム317のメンバーとして参加してきました.317のメンバーは,nojima, seikichi, qwerty の3人で,3人ともこの1年間ほとんど問題を解いていなかったので,ICPCでまともに戦えるか不安でしたが,まあ楽しめればいいやという軽い気持ちで参加しました.
以下,タイムライン.
- 開始後0分: 僕がテンプレートを打ち込んでいる間に,他のメンバーがA問題を読む.
- 開始後2分: A問題を読み終えたらしく僕と交代してseikichiくんがコードを書き始める.僕はC問題を読む.
- 開始後8分: A問題AC.続いてseikichi,qwertyペアがBに取りかかる.
- 開始後17分: B問題AC. 僕がC問題のコーディングに移る.
- 開始後30分: C問題のコーディングを終えるも,「色を1から6じゃなくて0から5で持つことにしたのに,ターゲットの色だけデクリメントを忘れる」「出力にカウントできるパネルは(0,0)から連結なパネルだけなのに,全体をカウントしていた」という2つのバグを仕込んでしまっており,デバッグに時間がかかる.
- 開始後44分: C問題AC. すでにD問題の疑似コードを紙に書いていたseikichiくんとqwertyさんがD問題のコーディングに入る.同時に,僕はE問題を読み始める
- 開始後70分: E問題の解法を考え終わって暇になったので,D問題のデバッグに参加する.
- 開始後76分: D問題AC. 僕とqwertyさんがE問題のコーディングに入る.
- 開始後105分: E問題のコーディングが終わる.グループ数はちゃんと出てるが,予備力がちゃんと出てない.予備力関係な場所は4行ぐらいしかないので,デバッグは簡単そう.
- 開始後111分: E問題AC. 全員でG問題に取り組む.
- 開始後150分: チーム内に絶望感が蔓延する.
- 開始後172分: seikichiくんが「Rewrite買いに行っていい?」とか言い始める.
- 開始後180分: 終わり.
結果は,全体7位で,学内3位でした.無事,予選通過できたようでよかったです.
Affinity Propagation を書いてみた (2)
昨日のコードは damping=0.5 で実行してたんだけど、ここのFAQを読むと、dampingの値は0.9がおすすめとか書いてあったので、dampingの値を修正してもう一度試してみた。ついでにコードの整理したので再掲。
ap.h
#pragma once #include <vector> std::vector<int> affinityPropagation( FILE* input, int prefType = 1, double damping = 0.9, int maxit = 1000, int convit = 50 );
ap.cpp
// An Implementation of Affinity Propergation // See: Clustering by Passing Messages Between Data Points #include <cstdio> #include <cstdlib> #include <cmath> #include <vector> #include <algorithm> #include <cassert> #include "ap.h" using namespace std; namespace { struct Edge { int src; // index of source int dst; // index of destination double s; // similarity s(src, dst) double r; // responsibility r(src, dst) double a; // availability a(src, dst) Edge(int src, int dst, double s, double r, double a) : src(src), dst(dst), s(s), r(r), a(a) {} }; typedef vector<Edge*> Edges; struct Graph { int n; // the number of vertices Edges* outEdges; // array of out edges of corresponding vertices Edges* inEdges; // array of in edges of corresponding vertices }; // Build graph from sparse similarity matrix stored in COO format. // Input specification is following: // First line contains an integer standing for the size of the matrix. // Following lines each contain two integers and a real number standing for // the row index i, the column index j and the similarity s(i,j) respectively. // Input ends with an end-of-file. // Note that this function does not check any errors in the given input. // Parameter: // input: Input file handle. // prefType: // 1: use median of similarities as preference // 2: use minimum of similarities as preference // 3: use min - (max - min) of similarities as preference Graph* buildGraph(FILE* input, int prefType) { Graph* graph = new Graph; fscanf(input, "%d", &graph->n); graph->outEdges = new Edges[graph->n]; graph->inEdges = new Edges[graph->n]; // read similarity matrix int i, j; double s; vector<double> ss; while (fscanf(input, "%d%d%lf", &i, &j, &s) != EOF) { if (i == j) { continue; } // add small noise to avoid degeneracies s += (1e-16 * s + 1e-300) * (rand() / (RAND_MAX + 1.0)); // make new edge Edge* e = new Edge(i, j, s, 0, 0); graph->outEdges[i].push_back(e); graph->inEdges[j].push_back(e); // store similarities for preference calculation ss.push_back(s); } // calculate preferences double pref; if (prefType == 1) { sort(ss.begin(), ss.end()); int m = ss.size(); pref = (m % 2) ? ss[m/2] : (ss[m/2 - 1] + ss[m/2]) / 2.0; } else if (prefType == 2) { pref = *min_element(ss.begin(), ss.end()); } else if (prefType == 3) { double minValue = *min_element(ss.begin(), ss.end()); double maxValue = *max_element(ss.begin(), ss.end()); pref = 2*minValue - maxValue; } else { assert(false); // invalid prefType } for (int i = 0; i < graph->n; ++i) { Edge* e = new Edge(i, i, pref, 0, 0); graph->outEdges[i].push_back(e); graph->inEdges[i].push_back(e); } return graph; } void destroyGraph(Graph* g) { for (int i = 0; i < g->n; ++i) { for (size_t j = 0; j < g->outEdges[i].size(); ++j) { delete g->outEdges[i][j]; } } delete [] g->outEdges; delete [] g->inEdges; delete g; } inline void update(double& variable, double newValue, double damping) { variable = damping * variable + (1.0 - damping) * newValue; } void updateResponsibilities(Graph* graph, double damping) { for (int i = 0; i < graph->n; ++i) { Edges& edges = graph->outEdges[i]; int m = edges.size(); double max1 = -HUGE_VAL, max2 = -HUGE_VAL; double argmax1 = -1; for (int k = 0; k < m; ++k) { double v = edges[k]->s + edges[k]->a; if (v > max1) { swap(max1, v); argmax1 = k; } if (v > max2) { max2 = v; } } // update responsibilities for (int k = 0; k < m; ++k) { if (k != argmax1) { update(edges[k]->r, edges[k]->s - max1, damping); } else { update(edges[k]->r, edges[k]->s - max2, damping); } } } } void updateAvailabilities(Graph* graph, double damping) { for (int k = 0; k < graph->n; ++k) { Edges& edges = graph->inEdges[k]; int m = edges.size(); // calculate sum of positive responsibilities double sum = 0.0; for (int i = 0; i < m-1; ++i) { sum += max(0.0, edges[i]->r); } // calculate availabilities double rkk = edges[m-1]->r; for (int i = 0; i < m-1; ++i) { update(edges[i]->a, min(0.0, rkk + sum - max(0.0, edges[i]->r)), damping); } // calculate self-availability update(edges[m-1]->a, sum, damping); } } bool updateExamplars(Graph* graph, vector<int>& examplar) { bool changed = false; for (int i = 0; i < graph->n; ++i) { Edges& edges = graph->outEdges[i]; int m = edges.size(); double maxValue = -HUGE_VAL; int argmax = i; for (int k = 0; k < m; ++k) { double value = edges[k]->a + edges[k]->r; if (value > maxValue) { maxValue = value; argmax = edges[k]->dst; } } if (examplar[i] != argmax) { examplar[i] = argmax; changed = true; } } return changed; } } // Cluster data points with Affinity Propagation. // Parameters: // input: Input file which contains sparse similarity matrix. see buildGraph(). // prefType: Specify what kind of preference we use. see buildGraph(). // damping: The damping factor. (0.5 <= damping < 1.0) // maxit: The maximum number of iterations. // convit: Specify how many iterations this algorithm stops when examplars // did not change for. // Returns: // Array of examplars of corresponding data points. vector<int> affinityPropagation(FILE* input, int prefType, double damping, int maxit, int convit) { assert(0.499 < damping && damping < 1.0); Graph* graph = buildGraph(input, prefType); vector<int> examplar(graph->n, -1); for (int i = 0, nochange = 0; i < maxit && nochange < convit; ++i, ++nochange) { updateResponsibilities(graph, damping); updateAvailabilities(graph, damping); if (updateExamplars(graph, examplar)) { nochange = 0; } } destroyGraph(graph); return examplar; }
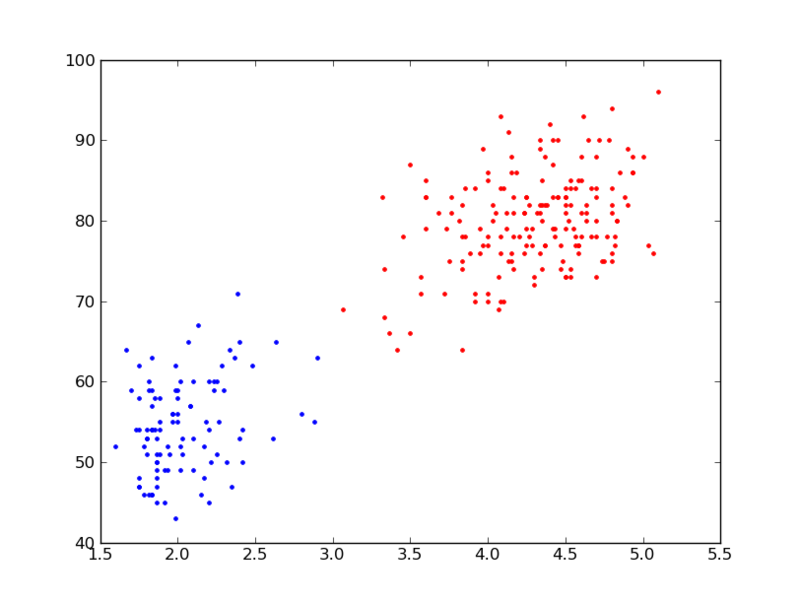
Old Faithfulに適用してみた結果。preference を類似度の最小値に設定した場合。昨日とちがってちゃんと2つに分かれてる。
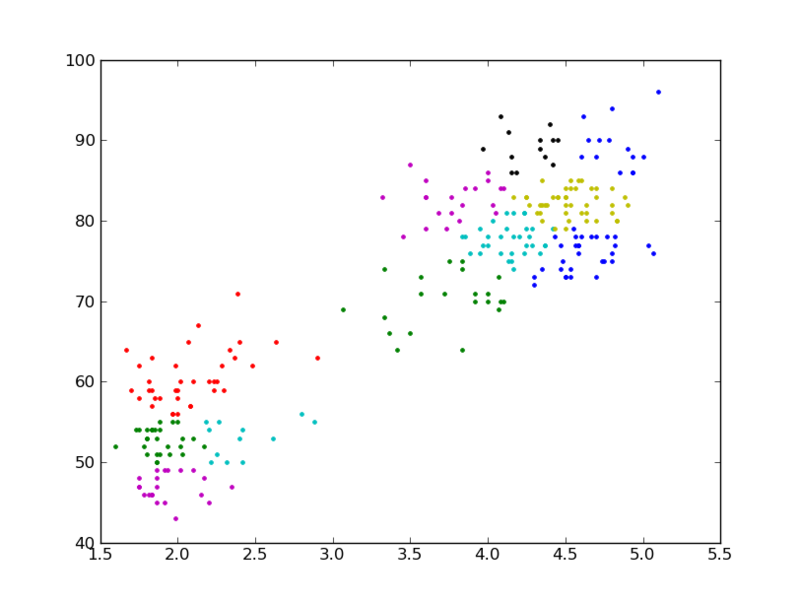
preferenceを類似度の中央値に設定すると多数のクラスタに分かれる。
Affinity Propagation を書いてみた
[追記] damping の値をあまり良くない値に設定してしまっていたらしいので、修正。→ http://d.hatena.ne.jp/nojima718/20101029/1288377507
Affinity Propagation を書いてみた.
おそらく自分でクラスタリングをするときは疎な空間でクラスタリングをすることになると思うので,疎な類似度に対して効率的にクラスタリングを行えるように,隣接リスト表現を使って Affinity Propagation を実装した.
計算量は,データ点の数をN,反復回数をTとして,グラフが密な場合は O(TN^2),疎な場合は O(TN).
(2010/10/30 修正 バグを見つけたのでコードは削除。 http://d.hatena.ne.jp/nojima718/20101029/1288377507 の方を見てください)
おなじみの Old faithful に対して 類似度=二乗誤差の-1倍 として適用してみた結果:
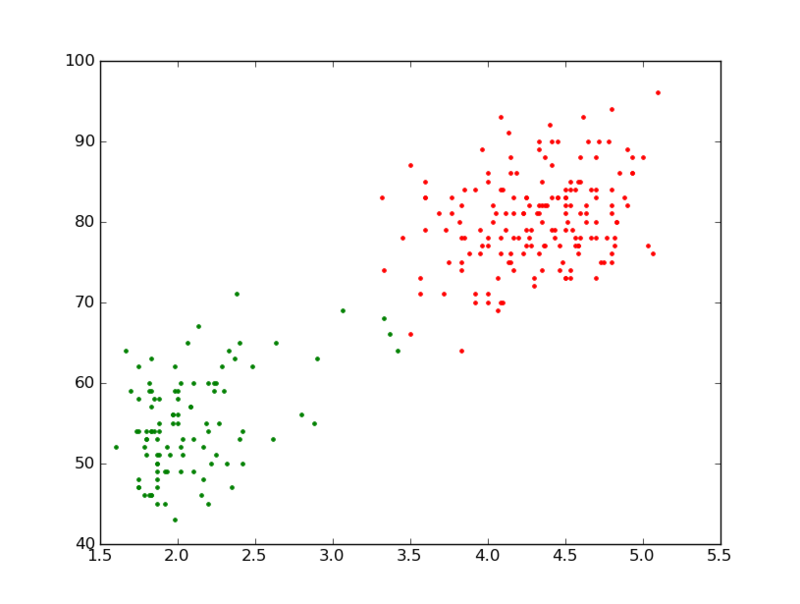
preference を類似度の最小値にした場合.3つのクラスタに分かれた.
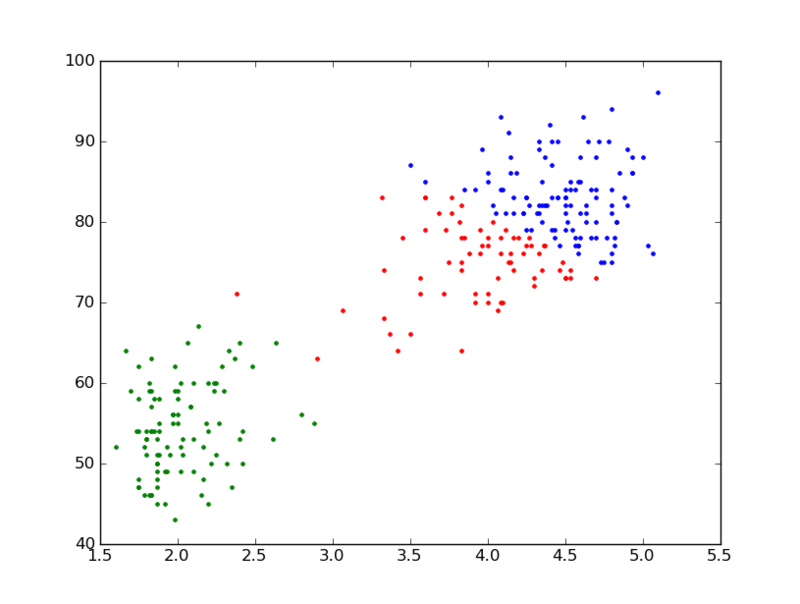
preference を類似度の最小値の1.2倍にした場合.2つのクラスタに分かれた.一番それっぽい.
preference を類似度の中央値にした場合.12個のクラスタに分かれた.
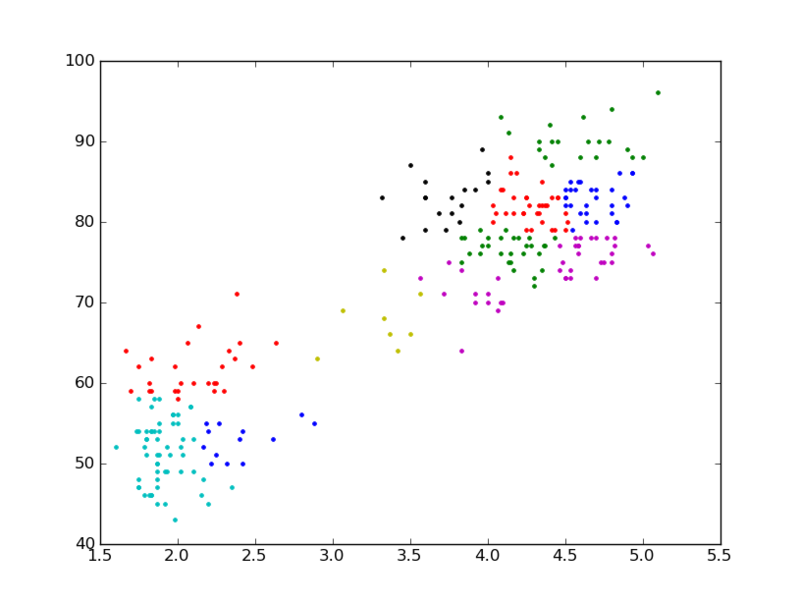
Affinity Propagation によるクラスタリング
Affinity Propagation というクラスタリングのアルゴリズムがあるらしいので調べてみた.
Affinity Propagation は,同じクラスタリングのアルゴリズムである k-means と比較すると次のようなメリットがある.
- 予めクラスタ数を決めておく必要がない.アルゴリズムが自動的にクラスタ数を決定する.
- 初期値依存性がない.k-means は,クラスタの中心点の初期値の選び方によって結果が変わる.
- 類似度 s(i,j) の制約が緩い.s(i,j) は,対称でなくてもいいし,三角不等式をみたしてなくてもよい.k-means は,可測であることを仮定している.
Affinity Propagation は,k-means と同じく,漸化式を反復計算していくアルゴリズムで,計算時間は,データ点の数をN,反復回数をTとして O(TN^2) となる.ただし,類似度 s(i,j) が疎である場合,すなわち s(i,j) > -∞ であるような s(i,j) が O(N) 個しかない場合は,O(TN) で計算できる.
アルゴリズムの内容については,日本語の解説を見つけたのでこっちを見ると良いかも.(最初は自分で書こうと思ってたけど既にあったので書く気がなくなった)